07重测序流程1-常规有参
〇. 一个项目出多份报告,看此节,否则往下看
## 在项目编号目录下,出几份报告就创建几个对应文件夹
1.显示$i文件夹
for i in `ls`;do cd $i && echo `ls` && cd -;done
2.查找染色体,默认选择前15个
for i in `ls`;do cd $i/data && pwd && ir.py -i *.fna -l | sort -k 1 -nr > len.txt && cat len.txt | head -n 15 | awk '{print $2}' > chrlist.txt && cat chrlist.txt && cd -;done
3.提取cds
for i in `ls`;do cd $i/data && gffread -x cds.fa -g *.fna *.gff && perl /share/nas6/zhouxy/functional_modules/cnvfmt_trans2longest/fasta2longest_with_gff3.pl -i cds.fa -g *.gff -o filter.cds.fa && cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/fungi_QAll.cfg ./ && cd - & done
4.填写filter.cds.fa绝对路径
## 真菌只改绝对路径
for i in `ls`;do cd $i/data && ll fungi_QAll.cfg && cd - & done
path=`pwd` && for i in `ls`; do echo "sed -i '1s|/share/nas6/zhanghk/project/ref/GP-20210708-3193_jiangnandaxue_12samples_jiaomu_trans/CBS7435/cbs7435_CDS.fa|"$path"/"$i"/data/filter.cds.fa|' "$path"/"$i"/data/fungi_QAll.cfg" >> modified1.sh;done && sh modified1.sh # 批量填写路径
5.功能注释
for i in `ls`;do cd $i/data && nohup perl /share/nas6/pub/pipline/gene-function-annotation/v1.3/Gene_Func_Anno_Pipline.pl --nr --swissprot --cog --kog --kegg --pfam --GO --cfg *_QAll.cfg --od Basic_function & done
6.生成配置文件
for i in `ls`;do cd $i && perl /share/nas6/xul/program/reseq/create_profile_for_reseq.pl -i 下发路径/2.clean_data/*/ -s data/*.fna -g data/chrlist.txt -gff data/*.gff -p $i -c data/Basic_function/Result/All_Database_annotation.xls && cd - & done
7.修改配置文件,运行主流程
for i in `ls`;do cd $i && nohup perl /share/nas1/yuj/pipline/reseq/v1.2/re_seq.pl -i ref_reseq_config.yaml -o analysis_dir & done
一.数据准备
主要是准备4.染色体列表,通常其他信息都有。
查找基因组看“8️⃣.查找参考基因组”
1. 测序所在路径,一般有多个文件夹,每个样品一个文件夹
2. 基因组fasta文件,有时需要下载,有时客户提供,还有常见的或者我们做过的可以到 /share/nas6/pub/genome中找找
gzip -d file.gz
gunzip file.gz
3. 基因组注释文件,gff3格式
## gbk转gff
perl /share/nas6/xul/program/chloroplast/mvista/genbank.to.gff.pl -i *.gbk -f *.fasta -o tmp.gff
perl -lane '/Name=(.*;)/;print $_."Parent=$1ID=$1"' tmp.gff > genome.gff #修改名字
4. 染色体列表,txt文件,一列
存放序列名称,因为有的fasta文件中有很多序列,该文件用于定义某些图片中需要展示的序列。
一般是保留染色体级别的序列,可以保留所有染色体
如果该基因组都是scaffold,那么就保留几个比较长的序列名称,十个左右就行
ir.py -i *.fna -l | sort -k 1 -nr > len.txt
cat len.txt | head -n 15 | awk '{print $2}' > chrlist.txt && cat chrlist.txt # 根据情况取前多少行
5. 功能注释结果的整合文件
二.功能注释 --CDS版
2.0 合并步骤
data]$
1.挑选cds
gffread -x cds.fa -g *.fna *.gff && perl /share/nas1/yuj/functional_modules/cnvfmt_trans2longest/fasta2longest_with_gff3.pl -i cds.fa -g *.gff -o filter.cds.fa
2.填写配置
## 真菌
cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/fungi_QAll.cfg ./
path=`pwd` && sed -i "1s|/share/nas6/zhanghk/project/ref/GP-20210708-3193_jiangnandaxue_12samples_jiaomu_trans/CBS7435/cbs7435_CDS.fa|$path/filter.cds.fa|" $path/fungi_QAll.cfg
## 细菌
cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/BCT_QAll.cfg ./
path=`pwd` && sed -i "1s|/share/nas6/yangl/project/2022/drna_seq/GP-20220113-3957_neinongda_2samples_xujun_trans/genomic/sequence.cds.fa|$path/filter.cds.fa|" $path/BCT_QAll.cfg
## 双子叶植物
cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/plant_QAll.cfg ./
path=`pwd` && sed -i "1s|/share/nas6/zhail/project/denoref/202106/GP-20210423-2913_shenyangnongye_fengxin_6samples_juhua_trans/analysis_dir/trinity_out_dir/Unigenes.fasta|$path/filter.cds.fa|" $path/plant_QAll.cfg
All_QAll.cfg
BCT_QAll.cfg #细菌只改绝对路径
danziye_plant_QAll.cfg
fungi_QAll.cfg #真菌只改绝对路径
Gene_Func_Anno_Pipline.cfg
INV_QAll.cfg
MAM_QAll.cfg
plant_QAll.cfg #植物改“绝对路径”“NR”“KEGG”,双子叶植物项目只改路径
ROD_QAll.cfg
VRL_QAll.cfg
VRT_QAll.cfg
3.功能注释
nohup perl /share/nas6/pub/pipline/gene-function-annotation/v1.3/Gene_Func_Anno_Pipline.pl --nr --swissprot --cog --kog --kegg --pfam --GO --cfg *_QAll.cfg --od Basic_function &
2.1 获取转录本序列文件
1. 获取转录本序列文件,fasta格式的核酸序列
$ gffread -x cds.fa -g *.fna *.gff
# 遇到错误就删删删 如下
$ grep -n "rna-A564_p001" GCF_004118075.2_ASM411807v2_genomic.gff | wc -l
$ for i in {1..6};do echo $i;sed -i '649514d' GCF_004118075.2_ASM411807v2_genomic.gff;done
# cds要用id来命名,重测序这块分析可能不用管
2.2 获取最长转录本
2.获取最长转录本
有的基因可能存在多个转录本,所以需要过滤下,根据上述提取出来的基因名称判断哪些是同一个基因的,一般id后面有.1 .2这种,就属于一个基因的不同转录本。
具体还要看提取出来的格式。
# perl /share/nas6/zhouxy/functional_modules/cnvfmt_trans2longest/fasta2longest.pl -i cds.fa -o filter.cds.fa # 旧的程序不用 不使用gff,不会改变序列id
$ perl /share/nas6/zhouxy/functional_modules/cnvfmt_trans2longest/fasta2longest_with_gff3.pl -i cds.fa -g *.gff -o filter.cds.fa # 用这个 会把rna的id替换成所属gene的id
$ perl /share/nas1/yuj/functional_modules/cnvfmt_trans2longest/fasta2longest_with_gff3.pl -i cds.fa -g *.gff -o filter.cds.fa # 输出id映射,后面富集分析记得改id
# perl /share/nas6/zhouxy/functional_modules/cnvfmt_trans2longest/fasta_trans_to_geneid_format_convert_ncbi.pl 格式转换 没用过
2.3 填写功能注释的配置文件
3. 填写功能注释的配置文件
## 植物 下面的nr库需要根据情况改成单子叶的或者双子叶的,默认双子叶
$ cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/plant_QAll.cfg ./
## 病毒
$ cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/VRL_QAll.cfg ./
##真菌只改绝对路径
$ cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/fungi_QAll.cfg ./
path=`pwd` && sed -i "1s|/share/nas6/zhanghk/project/ref/GP-20210708-3193_jiangnandaxue_12samples_jiaomu_trans/CBS7435/cbs7435_CDS.fa|$path/filter.cds.fa|" $path/fungi_QAll.cfg
修改第一行,改成filter.cds.fa的绝对路径!
2.4 运行功能注释
4. 运行功能注释
nohup perl /share/nas6/pub/pipline/gene-function-annotation/v1.3/Gene_Func_Anno_Pipline.pl --nr --swissprot --cog --kog --kegg --pfam --GO --cfg *_QAll.cfg --od Basic_function &
程序运行完后需要用到的文件就是:Basic_function/Result/All_Database_annotation.xls 这个文件,用于填写下面的配置文件
2.5 整合程序未运行的cog注释
## cog注释
perl /share/nas6/pub/pipline/gene-function-annotation/v1.3/bin/Gene_Cog_Anno.pl -id /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/202309-groupD/data/Basic_function/mid -Database /share/nas6/pub/database/cog/v20180610/myva -od /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/202309-groupD/data/Basic_function -Blast_cpu 80 -Blast_e 1e-5
## 整合进最终的xls文件
perl /share/nas6/pub/pipline/gene-function-annotation/v1.3/bin/anno_integrate.pl -gene /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/202309-groupD/data/filter.cds.fa -id /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/202309-groupD/data/Basic_function/Result -od /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/202309-groupD/data/Basic_function/Result -key filter.cds.fa
二.功能注释 -- 蛋白版
data]$
1.挑选cds
gffread -x cds.fa -g *.fna *.gff && perl /share/nas1/yuj/functional_modules/cnvfmt_trans2longest/fasta2longest_with_gff3.pl -i cds.fa -g *.gff -o filter.cds.fa && ir.py -s7 -n 1 -i filter.cds.fa -o filter.cds.pep
2.填写配置
第一行mrna用不到,不用管
## 细菌
cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/BCT_QAll.cfg ./
## 真菌
cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/fungi_QAll.cfg ./
## 双子叶
cp /share/nas6/pub/pipline/gene-function-annotation/v1.3/profile/plant_QAll.cfg ./
All_QAll.cfg
BCT_QAll.cfg
danziye_plant_QAll.cfg
fungi_QAll.cfg
Gene_Func_Anno_Pipline.cfg
INV_QAll.cfg
MAM_QAll.cfg
plant_QAll.cfg # 单子叶改NR/KEGG数据库,双子叶不用改
ROD_QAll.cfg
VRL_QAll.cfg
VRT_QAll.cfg
3.功能注释
nohup perl /share/nas6/pub/pipline/prot-function-annotation/current/Gene_Func_Anno_Pipline_v2.pl --all --cfg *_QAll.cfg --od Basic_function -fa filter.cds.pep &
三.有参流程
3.1 配置文件(v1.3流程需要分组文件)
配置模板:
/share/nas6/zhouxy/project/reseq/GP-20210820-3319_jiangsunongkeyuannongchanpinjiagongsuo_1samples_xijun/re_config.yaml
perl /share/nas6/xul/program/reseq/create_profile_for_reseq.pl
-i <in dir> contain all.gz file or fq or fastq
# 输入包含测序文件所在的路径(文件夹,不是具体文件路径),如 path/clean/*/
-s ref genome
# 输入参考基因组fasta文件
-g ref chr list
# 输入染色体列表
-c ref anno Gene_Anno/Result/All_Database_annotation.xls default
# 输入功能注释的结果Basic_function/Result/All_Database_annotation.xls,如果功能注释没做完,这个可以先不填。
-gff ref gff
# 输入gff文件
-Is insert_size,default 100-500
# 插入片段大小,一般不用改
-p Prefix default XM
# 物种名简拼,不改也行
cd ../
datapath=下发路径
for i in `ls $datapath/2.clean_data | grep -v txt`;do echo -e "$i\tALL";done > sample.group.txt
perl /share/nas6/xul/program/reseq/create_profile_for_reseq.pl -s data/*.fna -g data/chr*.* -gff data/*.gff* -c data/*/Result/All_Database_annotation.xls -p BY -i $datapath/2.clean_data/*/
# -i可以只输入制定样品 -i 2.clean_data/D1110/
echo -e "\n\tGroups : $(realpath sample.group.txt)" >> ref_reseq_config.yaml
3.2 运行主程序
原版/share/nas6/zhouxy/pipline/reseq/v1.3/re_seq.pl
## 不用了
# nohup perl /share/nas1/yuj/pipline/reseq/v1.2/re_seq.pl -i ref_reseq_config.yaml -o analysis_dir &
# 1.修改了循环数 2.bwa升级到bwa-mem2
# /share/nas1/yuj/pipline/reseq/v1.2/script/05vcf_stat/count_deg_indel.pl与原版有点区别,但效果应该一样
nohup perl /share/nas1/yuj/pipline/reseq/v1.3/re_seq.pl -i ref_reseq_config.yaml -o analysis_dir &
3.3 整理结果
# perl /share/nas1/yuj/pipline/reseq/v1.2/script/resultdir/resultDir.pl -i analysis_dir -o complete_dir &
# 去除了06里的cmds
# sh analysis_dir/.cmds_dir/genomeStat.cmds 有时候统计信息没生成
perl /share/nas1/yuj/pipline/reseq/v1.3/script/resultdir/resultDir.pl -i analysis_dir -o complete_dir &
3.4 生成报告
1.修改配置文件
# /share/nas6/zhouxy/pipline/reseq/current/re-report_html.cfg
cp /share/nas2/yuj/project/2024/re-sequencing/GP-20231007-6973_20240227/3/report.cfg ./report.cfg && realpath ./report.cfg
2.生成报告
# perl /share/nas6/zhouxy/pipline/reseq/current/re-report_html_for_lumpy_sv.pl -id complete_dir/ -cfg ./report.cfg # 有点小问题,先不用
# 售后QQ:2893941692
# perl /share/nas1/yuj/pipline/reseq/v1.2/re-report_html_for_lumpy_sv.pl -id complete_dir/ -cfg ./report.cfg
perl /share/nas1/yuj/pipline/reseq/v1.3/re-report_html_for_lumpy_sv.pl -id complete_dir/ -cfg ./report.cfg
-- 20230928 当基因功能注释文件夹不存在时去除报告相关内容
-- 20240426 当染色体结构变异文件夹不存在时去除报告相关内容
四.没有GFF时
舍弃一系列的indel部分
去掉某一步的rscript画图(可能不需要)
1.以下indel部分无法生成,会被过滤掉,跳过即可
touch analysis_dir/.flag_dir/gatk_VQSR.ok
touch analysis_dir/.flag_dir/gatk_VariantFiltration.ok
继续跑
2.跳过6.1 snpeff_index.ok这一步
可以不用管
3.在下一步注释,直接复制就行
cp analysis_dir/variation_dir/variants_anno_dir/samples.gatk.con.snp.vqsr.filter.vcf analysis_dir/variation_dir/variants_anno_dir/samples.gatk.con.snp.vqsr.filter.anno.vcf
4.运行Variant_Annotation.cmds的第2/3条命令
5.gatk_MergeVcfs.cmds去掉indel合并后运行
sh analysis_dir/.cmds_dir/gatk_MergeVcfs.cmds
touch analysis_dir/.flag_dir/gatk_MergeVcfs.ok
继续跑
6.diff_Variation.cmds去掉indel部分后运行
sh analysis_dir/.cmds_dir/diff_Variation.cmds
touch analysis_dir/.flag_dir/diff_Variation.ok
继续跑
7.analysis_dir/.cmds_dir/vcf_filter.cmds也会报错
不用管,能生成ok文件
8.popSNP_Stat.cmds去掉indel部分后运行
sh analysis_dir/.cmds_dir/popSNP_Stat.cmds
touch analysis_dir/.flag_dir/popSNP_Stat.ok
继续跑
五.出现的问题(v1.3)
5.1 SV无结果
ll analysis_dir/SV/groups_detect # 里面是空文件
重新运行即可
sh analysis_dir/.cmds_dir/all.SVdetect.cmds &
5.2 mapping_dir图片(真菌项目)
5.2.1 bam2depth
1.bam2depth.cmds
depth文件大小差别大,是因为没有输出0位置
samtools depth -m 100000 -a # 最大值100000,输出0位置
touch analysis_dir/.flag_dir/bam2depth.ok
5.2.2 深度窗口绘图
2.depth2windows.cmds
真菌染色体短,窗口设置成1k
-d 1000
touch analysis_dir/.flag_dir/depth2windows.ok
3.绘制轨道图(注意深度)
sh analysis_dir/.cmds_dir/depth_Track_point.cmds
touch analysis_dir/.flag_dir/depth_Track_point.ok
5.2.3 mapping_stat/bcparse
3.bam2bc_stat.cmds samples_mapping_stat.cmds
见v1.2的5.2.1
先生成bc,再重新绘图
for i in {BY4741,FG2-QC,XNB};do /share/nas1/yuj/pipline/reseq/v1.3/script/01bam/coverage_depth.R --i $i.coverage.csv --o $i.coverage;done
5.3 snpeff运行缓慢 Variant_Annotation.cmds
换一个版本来跑这一步
/share/nas6/zhouxy/software/java/current/bin/java -jar /share/nas6/yangl/biosoft/snpEff/snpEff_v3_6/snpEff.jar MH -o gatk -ud 2000 -csvStats -s /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.snp.stat.csv -s /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.snp.stat.html -c /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/snpeff_index/MH.config -v /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.gatk.con.snp.vqsr.filter.vcf > /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.gatk.con.snp.vqsr.filter.anno.vcf
/share/nas6/zhouxy/biosoft/perl/current/bin/perl /share/nas1/yuj/pipline/reseq/v1.3/script/02variant/extract_oneEff_anno.pl -i /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.gatk.con.snp.vqsr.filter.anno.vcf -o /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.pop.snp.anno.result.vcf
/share/nas6/zhouxy/biosoft/perl/current/bin/perl /share/nas1/yuj/pipline/reseq/v1.3/script/02variant/vcf_to_snplist_v1.4.pl -i /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.pop.snp.anno.result.vcf -o /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.pop.snp.anno.result.list -ref 1
## indel
/share/nas6/zhouxy/software/java/current/bin/java -jar /share/nas6/yangl/biosoft/snpEff/snpEff_v3_6/snpEff.jar MH -o gatk -ud 2000 -csvStats -s -s/share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.indel.stat.csv -s /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.indel.stat.html -c /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/snpeff_index/MH.config -v /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.gatk.con.indel.vqsr.filter.vcf > /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.gatk.con.indel.vqsr.filter.anno.vcf
/share/nas6/zhouxy/biosoft/perl/current/bin/perl /share/nas1/yuj/pipline/reseq/v1.3/script/02variant/extract_oneEff_anno.pl -i /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.gatk.con.indel.vqsr.filter.anno.vcf -o /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.pop.indel.anno.result.vcf
/share/nas6/zhouxy/biosoft/perl/current/bin/perl /share/nas1/yuj/pipline/reseq/v1.3/script/02variant/vcf_to_indellist_v1.5.pl -i /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.pop.indel.anno.result.vcf -o /share/nas2/yuj/project/2025/re-sequencing/GP-20241021-9521-2_20250104/analysis_dir/variation_dir/variants_anno_dir/samples.pop.indel.anno.result.list -ref 1
五.检查结果(v1.2)
5.1 SV无结果
ll analysis_dir/SV/groups_detect # 里面是空文件
重新运行即可
sh analysis_dir/.cmds_dir/all.SVdetect.cmds &
5.2 bcparse 测序深度/插入片段
5.2.0 重新运行
通常重新运行samples_align_stats.cmds即可
但有时候这个cmd文件未生成,
需要运行下面命令
perl /share/nas1/yuj/pipline/reseq/v1.2/script/01bam/samtools_stats_parser2.pl -i analysis_dir/mapping_dir/mapping_stat/bcfiles -o analysis_dir/mapping_dir/mapping_stat/bcparse -r analysis_dir/genome/*.fna
display analysis_dir/mapping_dir/mapping_stat/bcparse/*.coverage.png
display analysis_dir/mapping_dir/mapping_stat/bcparse/*.insertsize.png
5.2.1 Q1:测序深度太高
1.
cd analysis_dir/mapping_dir/mapping_stat/bcparse
2.根据*.coverage.csv深度来调整else分支的最大值,其控制的是下图中的横坐标最大值
/share/nas1/yuj/pipline/reseq/v1.2/script/01bam/coverage_depth.R 42行
if (xmean < 10){xmax=20;}else{xmax=200} # 默认为200,深度高就改成1000
3.然后先尝试一次画图
/share/nas1/yuj/pipline/reseq/v1.2/script/01bam/coverage_depth.R --i *.coverage.csv --o $sample.coverage
4.不行的话再看下面从头调整文件然后画图
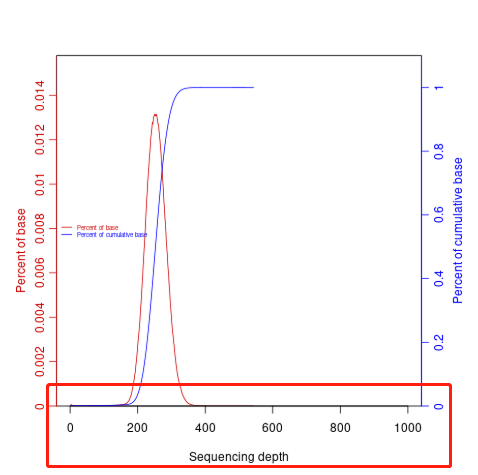
参数说明:
-c, --coverage <int>,<int>,<int>:覆盖度分布的最小值、最大值和步长,默认为1,1000,1
1)遇到细菌项目,可以 -c 1,10000000,1
然后根据实际情况再去更改上图的xmax
2)以下都是某个真菌项目,更改的是步长参数,xmax参数未改
## 单个样品for循环 step从100画到2900
analysis_dir/mapping_dir/01.bwa2sortbam]$
for i in {1..29};do samtools stats -c 1,50000,$i"00" EFV1.sort.bam > ../mapping_stat/bcfiles/$i"00EFV1.bc" && grep ^COV ../mapping_stat/bcfiles/$i"00EFV1.bc" | cut -f 3- > ../mapping_stat/bcparse/$i"00EFV1.coverage.csv" && cd ../mapping_stat/bcparse/ && /share/nas1/yuj/pipline/reseq/v1.2/script/01bam/coverage_depth.R --i $i"00EFV1.coverage.csv" --o $i"00EFV1.coverage" && cd - & done
## 固定参数 for循环 挨个样品画
for i in {Del-E,Del-L,FAdV-4,H9-HA-E,H9-HA-L,rF2}; do samtools stats -c 1,50000,1500 $i.sort.bam | grep ^COV | cut -f 3- > ../mapping_stat/bcparse/$i.coverage.csv && cd ../mapping_stat/bcparse/ && /share/nas1/yuj/pipline/reseq/v1.2/script/01bam/coverage_depth.R --i $i.coverage.csv --o $i.coverage && cd - ;done
## 单样品画图 手动修改参数
samtools stats -c 1,500000,1 EFV1.sort.bam > ../mapping_stat/bcfiles/EFV1.bc && grep ^COV ../mapping_stat/bcfiles/EFV1.bc | cut -f 3- > ../mapping_stat/bcparse/EFV1.coverage.csv && cd ../mapping_stat/bcparse/ && /share/nas1/yuj/pipline/reseq/v1.2/script/01bam/coverage_depth.R --i EFV1.coverage.csv --o EFV1.coverage && cd -
5.2.2 Q2:插入片段重影
1.R
$ Rscript /share/nas1/yuj/pipline/reseq/v1.2/script/01bam/insert_size.R --i I4.insertsize.csv --o I4.insertsize # 原流程存在重影问题
2.python
python3 /share/nas1/yuj/pipline/reseq/v1.2/script/01bam/insert_size.R.py -i *.insertsize.csv -o $sample.insertsize # 修复了重影问题
5.3 富集分析没有图
display analysis_dir/variant_gene/snp.variant_gene_Annotation/GO/snp.variant_gene.GO.png
0.做熟悉后,发现缺gene-前缀可以直接看这一步,下面其他步骤就不用看了
一股脑全部运行就行
perl /share/nas1/yuj/pipline/reseq/v1.2/script/05vcf_stat/region_info_filterd.pl -i data/Basic_function/Result/All_Database_annotation.xls -s analysis_dir/vcf_filter/samples.pop.snp.filter.list -o analysis_dir/vcf_filter/samples.pop.snp.filter.anno.xls &
perl /share/nas1/yuj/pipline/reseq/v1.2/script/05vcf_stat/region_info_filterd.pl -i data/Basic_function/Result/All_Database_annotation.xls -s analysis_dir/vcf_filter/samples.pop.indel.filter.list -o analysis_dir/vcf_filter/samples.pop.indel.filter.anno.xls &
tmp=`sed -n "2p" analysis_dir/variant_gene/snp.variant_gene.ID | awk -F "_" '{print $1}'` && sed -i "s/$tmp/gene-$tmp/g" analysis_dir/variant_gene/snp.variant_gene.ID && sed -i "s/$tmp/gene-$tmp/g" analysis_dir/variant_gene/indel.variant_gene.ID
sh /share/nas1/yuj/pipline/reseq/v1.2/script/07DEG_Anno/DEG_Anno.sh analysis_dir/variant_gene/snp.variant_gene.ID snp.variant_gene analysis_dir/variant_gene data/Basic_function/Result/All_Database_annotation.xls && perl /share/nas1/yuj/pipline/reseq/v1.2/script/07DEG_Anno/anno.stat.pl -i analysis_dir/variant_gene -o analysis_dir/variant_gene/anno.stat.xls &
sh /share/nas1/yuj/pipline/reseq/v1.2/script/07DEG_Anno/DEG_Anno.sh analysis_dir/variant_gene/indel.variant_gene.ID indel.variant_gene analysis_dir/variant_gene data/Basic_function/Result/All_Database_annotation.xls && perl /share/nas1/yuj/pipline/reseq/v1.2/script/07DEG_Anno/anno.stat.pl -i analysis_dir/variant_gene -o analysis_dir/variant_gene/anno.stat.xls &
sh /share/nas1/yuj/pipline/reseq/v1.2/script/variant_gene.sh # 上面所有命令都写入了该文件
1.vcf_filter.cmds里面snp/indel的最后一部分重新运行
/share/nas6/zhouxy/biosoft/perl/current/bin/perl /share/nas1/yuj/pipline/reseq/v1.2/script/05vcf_stat/region_info_filterd.pl
放后台就行
perl /share/nas1/yuj/pipline/reseq/v1.2/script/05vcf_stat/region_info_filterd.pl -i data/Basic_function/Result/All_Database_annotation.xls -s analysis_dir/vcf_filter/samples.pop.snp.filter.list -o analysis_dir/vcf_filter/samples.pop.snp.filter.anno.xls &
perl /share/nas1/yuj/pipline/reseq/v1.2/script/05vcf_stat/region_info_filterd.pl -i data/Basic_function/Result/All_Database_annotation.xls -s analysis_dir/vcf_filter/samples.pop.indel.filter.list -o analysis_dir/vcf_filter/samples.pop.indel.filter.anno.xls &
2.生成mapping.id
cd data && perl /share/nas1/yuj/functional_modules/cnvfmt_trans2longest/fasta2longest_with_gff3.pl -i cds.fa -g *.gff -o tmp && cp mapping.id ../analysis_dir/variant_gene/ && cd ../analysis_dir/variant_gene/ && cd ../../ # 输出id映射
3.variant_gene.cmds里面运行snp/indel第一部分生成ID文件
perl /share/nas1/yuj/pipline/reseq/v1.2/script/06varGene/get_gene_info.pl -i analysis_dir/vcf_filter/samples.pop.snp.filter.list -o analysis_dir/variant_gene/snp.variant_gene.ID &
perl /share/nas1/yuj/pipline/reseq/v1.2/script/06varGene/get_gene_info.pl -i analysis_dir/vcf_filter/samples.pop.indel.filter.list -o analysis_dir/variant_gene/indel.variant_gene.ID &
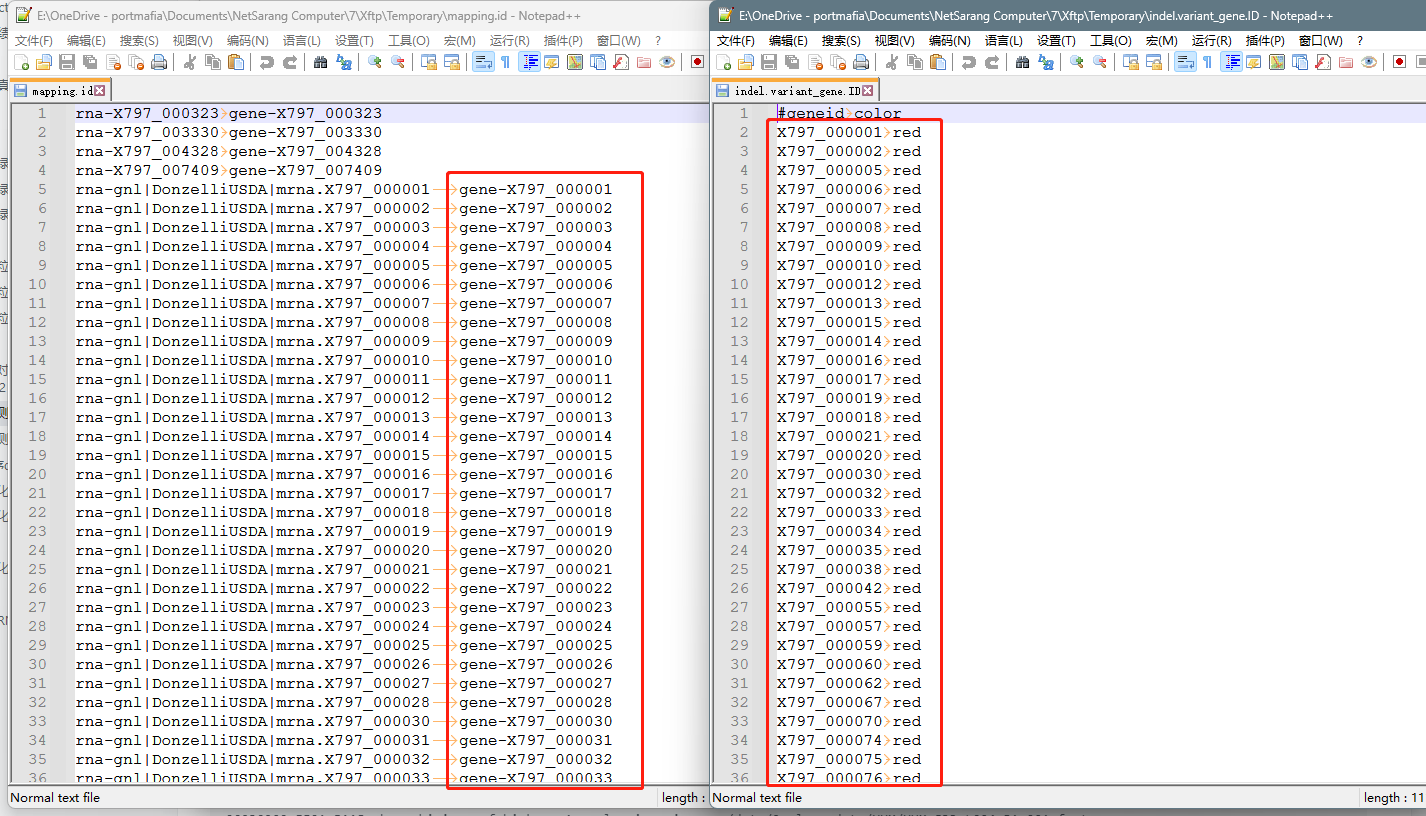
4.修改ID文件里的前缀,再运行替换命令
4.1 如果差别仅仅只是gene-,如上图所示,右侧框里少了gene-,不用awk来替换,直接文本编辑就ok,这一步的命令可以跳过
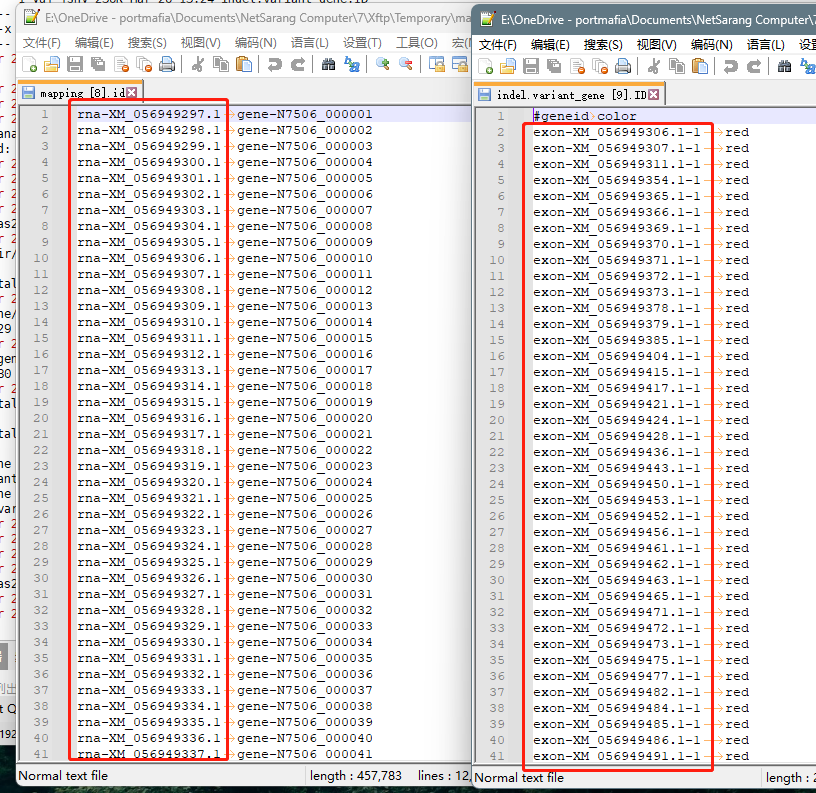
4.2 但如果是上图所示,就需要手动先把右侧文件编辑成左边框的格式,然后运行下面的命令
# 注意分隔用制表符
analysis_dir/variant_gene]
$ awk 'BEGIN{OFS="\t"} NR==FNR{mapping[$1]=$2; next} $1 in mapping{$1=mapping[$1]} 1' mapping.id indel.variant_gene.ID > indel.variant_gene.ID2
$ awk 'BEGIN{OFS="\t"} NR==FNR{mapping[$1]=$2; next} $1 in mapping{$1=mapping[$1]} 1' mapping.id snp.variant_gene.ID > snp.variant_gene.ID2
$ rm *.ID mapping.id && rename ID2 ID *
5.variant_gene.cmds里面snp/indel从sh开始运行
sh /share/nas1/yuj/pipline/reseq/v1.2/script/07DEG_Anno/DEG_Anno.sh analysis_dir/variant_gene/snp.variant_gene.ID snp.variant_gene analysis_dir/variant_gene data/Basic_function/Result/All_Database_annotation.xls && perl /share/nas1/yuj/pipline/reseq/v1.2/script/07DEG_Anno/anno.stat.pl -i analysis_dir/variant_gene -o analysis_dir/variant_gene/anno.stat.xls &
sh /share/nas1/yuj/pipline/reseq/v1.2/script/07DEG_Anno/DEG_Anno.sh analysis_dir/variant_gene/indel.variant_gene.ID indel.variant_gene analysis_dir/variant_gene data/Basic_function/Result/All_Database_annotation.xls && perl /share/nas1/yuj/pipline/reseq/v1.2/script/07DEG_Anno/anno.stat.pl -i analysis_dir/variant_gene -o analysis_dir/variant_gene/anno.stat.xls &
5.4 密度图
针对几万bp长度的染色体
/share/nas1/yuj/pipline/reseq/v1.2/script/03vcf/VariationDraw_kb.pl -b 1k
5.4 mapping_depth图片未生成 通常不管
会提示id不在输入文件进而无法绘图
另外,发现图形不是平滑曲线,也是参数的问题
换参数重新生成fordraw可以解决
重新运行
## 程序
1.滑窗统计
/share/nas6/zhouxy/biosoft/bin/depth_stat_windows
-i file input file
-o file output file
-w num windows size
-h usage
-w参数默认使用1000000,生成的fordraw如下,第二列都是1
JAKREL010000100.1 1 10.531282
JAKREL010000101.1 1 9.961758
JAKREL010000102.1 1 10.662214
JAKREL010000103.1 1 10.242439
JAKREL010000104.1 1 9.998680
JAKREL010000105.1 1 10.267645
JAKREL010000106.1 1 9.822779
2.r绘图
/share/nas1/yuj/pipline/reseq/v1.2/script/01bam/genomeCoveragehorizontalArea.R
上个程序 -w 1000000 对应绘图程序的 -unit Mb
以真菌如何修改为例(真菌染色体长度较短,导致参数不匹配)
1.修改bam2depth.cmds第二部分的 -w参数值
根据len.txt中最长长度135547,-w设置为1000,也就是以Kb为单位
修改成如下/share/nas6/zhouxy/biosoft/samtools/current/bin/samtools depth /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/m8/analysis_dir/mapping_dir/01.bwa2sortbam/M8.sort.bam > /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/m8/analysis_dir/mapping_dir/mapping_depth/M8.depth && /share/nas6/zhouxy/biosoft/bin/depth_stat_windows -i /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/m8/analysis_dir/mapping_dir/mapping_depth/M8.depth -o /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/m8/analysis_dir/mapping_dir/mapping_depth/M8.depth.fordraw -w 1000
head data/len.txt && sed -i 's/-w 1000000/-w 1000/g' analysis_dir/.cmds_dir/bam2depth.cmds && sh analysis_dir/.cmds_dir/bam2depth.cmds &
2.genomeCoveragehorizontalArea.cmds修改 --unit为Kb
修改成如下/share/nas6/zhouxy/biosoft/R/current/bin/Rscript /share/nas1/yuj/pipline/reseq/v1.2/script/01bam/genomeCoveragehorizontalArea.R --infile /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/m8/analysis_dir/mapping_dir/mapping_depth/M8.depth.fordraw --idfile /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/m8/data/chrlist.txt --outfile /share/nas1/yuj/project/re-sequencing/GP-20230327-5799_20230519/m8/analysis_dir/mapping_dir/mapping_depth/M8.genome.coverage --group.col 1 --x.col 2 --y.col 3 --x.lab Sequence-Position --y.lab AverageDepth-log2 --skip 0 --unit Kb --log2
sed -i 's/--unit Mb/--unit Kb/g' analysis_dir/.cmds_dir/genomeCoveragehorizontalArea.cmds && sh analysis_dir/.cmds_dir/genomeCoveragehorizontalArea.cmds &
5.5 samtools index: failed to create index for "xxx" Numerical result out of range
$ /share/nas1/yuj/software/bwa-mem2-2.2.1_x64-linux/bwa-mem2 mem -M -t 24 -R '@RG\tID:HHM\tLB:HHM\tPL:ILLUMINA\tSM:HHM' /share/nas1/yuj/project/re-sequencing/GP-20230209-5591_20230317/analysis_dir/genome/iwgsc_refseqv2.1_assembly.fa /share/nas1/seqloader/reseq/GP-20230209-5591_JAAS_zhongzhixiyuan_fubisheng_1sample_xiaomai_reseq/data/2.clean_data/HHM/HHM_S33_L004_R1_001.fastq.gz /share/nas1/seqloader/reseq/GP-20230209-5591_JAAS_zhongzhixiyuan_fubisheng_1sample_xiaomai_reseq/data/2.clean_data/HHM/HHM_S33_L004_R2_001.fastq.gz | /share/nas6/zhouxy/biosoft/samtools/current/bin/samtools sort -O bam -@ 4 -m 1G -T /share/nas1/yuj/project/re-sequencing/GP-20230209-5591_20230317/analysis_dir/mapping_dir/01.bwa2sortbam/HHM -o /share/nas1/yuj/project/re-sequencing/GP-20230209-5591_20230317/analysis_dir/mapping_dir/01.bwa2sortbam/HHM.sort.bam - && /share/nas6/zhouxy/biosoft/samtools/current/bin/samtools index /share/nas1/yuj/project/re-sequencing/GP-20230209-5591_20230317/analysis_dir/mapping_dir/01.bwa2sortbam/HHM.sort.bam &
报错信息如下:
[E::hts_idx_push] Region 536870840..536870990 cannot be stored in a bai index. Try using a csi index with min_shift = 14, n_lvls >= 6
samtools index: failed to create index for "/share/nas1/yuj/project/re-sequencing/GP-20230209-5591_20230317/analysis_dir/mapping_dir/01.bwa2sortbam/HHM.sort.bam": Numerical result out of range
解决方法:换用其他版本
$ wget https://sourceforge.net/projects/samtools/files/samtools/0.1.13/samtools-0.1.13.tar.bz2 --no-check-certificate
$ tar -jxvf samtools-0.1.13.tar.bz2
$ cd /share/nas1/yuj/software/samtools-0.1.13
$ make
$ ./samtools
$ /share/nas1/yuj/software/samtools-0.1.13/samtools index /share/nas1/yuj/project/re-sequencing/GP-20230209-5591_20230317/analysis_dir/mapping_dir/01.bwa2sortbam/HHM.sort.bam &
"samtools index failed - 简书." https://www.jianshu.com/p/75ad536ef47b, 访问时间 1 Jan. 1970.
5.6 位点太少(这个解决不太对,后面再整理)
02.bamdedup]
$ for i in *.bam;do echo $i;bcftools mpileup -Ou -f Fowl_aviadenovirus_4.fasta(参考基因组) $i | bcftools call -mv -Ov -o "output_"${i%.sort.dedup.bam}".vcf" & done
六.从vcf中挑选指定区间结果
6.0项目应用
1.list文件没有头信息 直接处理
$ vcftools --vcf samples.pop.snp.anno.result.list --chr Chr4A --from-bp 742765746 --to-bp 744656845 --recode --recode-INFO-all --out Chr4A.742765746-744656845.snp.anno.result.list # snp
$ vcftools --vcf samples.pop.indel.anno.result.list --chr Chr4A --from-bp 742765746 --to-bp 744656845 --recode --recode-INFO-all --out Chr4A.742765746-744656845.indel.anno.result.list # indel
2.vcf有head信息 直接处理,头信息会包含所有染色体
$ vcftools --gzvcf/--vcf samples.pop.snp.anno.result.vcf.gz/samples.pop.snp.anno.result.vcf --chr Chr4A --from-bp 742765746 --to-bp 744656845 --recode --recode-INFO-all --out Chr4A.742765746-744656845.snp.anno.result.vcf
3.对于vcf文件 需要进一步处理 头信息只包含所需染色体
$ mv all.sv.gt.anno.result.vcf unsorted.all.sv.gt.anno.result.vcf # sv
$ bcftools sort unsorted.all.sv.gt.anno.result.vcf -o all.sv.gt.anno.result.vcf # sv
$ cp samples.pop.snp.anno.result.vcf samples.pop.snp.anno.result.vcf.bak # snp
$ cp samples.pop.indel.anno.result.vcf samples.pop.indel.anno.result.vcf.bak # indel
$ cp all.sv.gt.anno.result.vcf all.sv.gt.anno.result.vcf.bak # sv
$ bgzip samples.pop.snp.anno.result.vcf # 压缩 snp
$ bgzip samples.pop.indel.anno.result.vcf # 压缩 indel
$ bgzip all.sv.gt.anno.result.vcf # 压缩 sv
$ bcftools index samples.pop.snp.anno.result.vcf.gz # 索引 snp
$ bcftools index samples.pop.indel.anno.result.vcf.gz # 索引 indel
$ bcftools index all.sv.gt.anno.result.vcf.gz # 索引 sv
$ (bcftools view -h samples.pop.snp.anno.result.vcf.gz | grep -v "##contig" | sed "/^##reference/a $(bcftools view -h samples.pop.snp.anno.result.vcf.gz | grep -w "ID=Chr4A")" ; bcftools view -H -r Chr4A:742765746-744656845 samples.pop.snp.anno.result.vcf.gz ) | bcftools view -Oz -o Chr4A.742765746-744656845.snp.anno.result.vcf.gz # 输出为.gz文件
$ (bcftools view -h samples.pop.snp.anno.result.vcf.gz | grep -v "##contig" | sed "/^##reference/a $(bcftools view -h samples.pop.snp.anno.result.vcf.gz | grep -w "ID=Chr4A")" ; bcftools view -H -r Chr4A:742765746-744656845 samples.pop.snp.anno.result.vcf.gz ) > Chr4A.742765746-744656845.snp.anno.result.vcf # 直接输出 snp
$ (bcftools view -h samples.pop.indel.anno.result.vcf.gz | grep -v "##contig" | sed "/^##reference/a $(bcftools view -h samples.pop.indel.anno.result.vcf.gz | grep -w "ID=Chr4A")" ; bcftools view -H -r Chr4A:742765746-744656845 samples.pop.indel.anno.result.vcf.gz ) > Chr4A.742765746-744656845.indel.anno.result.vcf # 直接输出 indel
$ (bcftools view -h all.sv.gt.anno.result.vcf.gz | grep -v "##contig" | sed "/^##reference/a $(bcftools view -h all.sv.gt.anno.result.vcf.gz | grep -w "ID=Chr4A")" ; bcftools view -H -r Chr4A:742765746-744656845 all.sv.gt.anno.result.vcf.gz ) > Chr4A.742765746-744656845.sv.gt.anno.result.vcf # 直接输出 sv
$ gunzip -c samples.pop.snp.anno.result.vcf.gz > samples.pop.snp.anno.result.vcf # 保留源文件 解压 snp
$ gunzip -c samples.pop.indel.anno.result.vcf.gz > samples.pop.indel.anno.result.vcf # 保留源文件 解压 indel
$ gunzip -c all.sv.gt.anno.result.vcf.gz > all.sv.gt.anno.result.vcf # 保留源文件 解压 sv
6.1vcftools不需要索引
# 不一定需要vcf 只要满足第一列染色体/第二列位置即可
$ vcftools --gzvcf samples.pop.snp.anno.result.vcf.gz --chr Chr4A --from-bp 742765746 --to-bp 744656845 --recode --recode-INFO-all --out Chr4A.742765746-744656845 # 测试ok
$ vcftools --vcf samples.pop.snp.anno.result.list --chr Chr4A --from-bp 742765746 --to-bp 744656845 --recode --recode-INFO-all --out Chr4A.742765746-744656845.list # 测试ok
vcftools --gzvcf input.vcf --chr n --recode – recode-INFO-all --stdout | gzip -c > output.vcf.gz
说明:
–gzvcf:处理压缩格式的vcf文件(可替换为–vcf)
–chr n:选择染色体n,例:–chr 1
–recode:重新编码为vcf文件,有过滤操作都要加上--recode
–recode-INFO-all:将输出的文件保存所有INFO信息
–stdout:标准输出,后接管道命令
–gzip -c:压缩
--max-missing
--max-missing的取值是0-1,为1时表示某个位点上所有的样本必须都有基因型,一个样本的基因型都不能缺。所以这个选项可以理解为:能分型的样本占总样本的比例至少为多少。
关联笔记 : ../群体遗传学/根据基因染色体坐标区间提取区间内SNP位点信息
6.2 bcftools需要tbi索引(!!用这个!!)
1.取染色体上某一区间
$ bcftools filter 1000Genomes.vcf.gz --regions 9:4700000-4800000 > 4700000-4800000.vcf # 未测试
$ bcftools view -r 1:100000-200000 input.vcf.gz -o output.vcf.gz # 未测试
2.大批量取位点
染色体及位置文件chr_pos.list,示例如下
chr1 27639
chr1 60383
chr2 60469
chr3 60516
chr4 60534
或者直接给区间也行
chr1 1 1000
chr1 2000 4500
bgzip samples.snp.region.filter.vcf && tabix -p vcf samples.snp.region.filter.vcf.gz && bcftools view -R chr_pos.list samples.snp.region.filter.vcf.gz > new.samples.snp.region.filter.vcf
ref:[bcftools 提取vcf(snp/indel)文件子集 - 天使不设防 - 博客园]
(https://www.cnblogs.com/mmtinfo/p/11945592.html)
6.3shell+bcftools需要建立索引
$ bcftools index samples.pop.snp.anno.result.vcf.gz # 索引
$ (bcftools view -h samples.pop.snp.anno.result.vcf.gz | grep -v "##contig" | sed "/^##reference/a $(bcftools view -h samples.pop.snp.anno.result.vcf.gz | grep -w "ID=Chr4A")" ; bcftools view -H samples.pop.snp.anno.result.vcf.gz Chr4A ) | bcftools view -Oz -o Chr4A.vcf.gz
这条命令使用了多个bcftools命令来处理名为samples.pop.snp.anno.result.vcf.gz的压缩VCF文件,并将结果保存为Chr4A.vcf.gz。
以下是对每个命令和整个命令的解释:
bcftools view -h samples.pop.snp.anno.result.vcf.gz: 这个命令将输出samples.pop.snp.anno.result.vcf.gz文件的头部信息,其中包含了关于变异数据的元数据。grep -v "##contig": 这个命令使用grep工具过滤掉头部信息中包含"##contig"的行,即排除了关于contig的信息。sed "/^##reference/a $(bcftools view -h samples.pop.snp.anno.result.vcf.gz | grep -w "ID=Chr4A")": 这个命令使用sed工具在头部信息的行中插入关于Chr4A的信息。它通过子命令$(bcftools view -h samples.pop.snp.anno.result.vcf.gz | grep -w "ID=Chr4A")获取关于Chr4A的元数据,并使用/^##reference/a将其插入到头部信息的合适位置。bcftools view -H samples.pop.snp.anno.result.vcf.gz Chr4A: 这个命令使用bcftools view从samples.pop.snp.anno.result.vcf.gz文件中提取Chr4A的变异数据,并丢弃头部信息。|: 这个管道符号将第3步和第4步的输出连接起来,作为下一个命令的输入。bcftools view -Oz -o Chr4A.vcf.gz: 这个命令将前面的结果通过bcftools view命令再次处理,并使用-Oz选项将输出压缩为.vcf.gz格式,并将结果保存为Chr4A.vcf.gz文件。
综合起来,该命令的目的是从一个压缩的VCF文件中提取Chr4A的变异数据,并将结果保存为压缩的VCF文件。
来源 : shell高级技巧:提取vcf文件中一个contig_徐洲更hoptop的博客-CSDN博客
# 可以把下面的函数添加到.bashrc中
# function
subvcf()
{
(bcftools view -h $1 | grep -v "##contig" | sed "/^##reference/a $(bcftools view -h $1| grep -w "ID=$2")" ; bcftools view -H $1 $2 ) | bcftools view -Oz -o $2.vcf.gz && bcftoos index $2.vcf.gz
}
# usage: subvcf vcf/bcf contig1/chr1
七.外源基因插入位点流程
140Bioinfo/重测序&简化测序/06T-DNA(外源基因)插入位点
八.查找参考基因组
## 根据登录号下载
ncbi-genome-download --assembly-accessions refseq.txt plant --section refseq --formats fasta,gff --progress-bar --parallel 3 -o data1 --flat-output
## 根据物种名
ncbi-genome-download --genera "Pseudomonas reinekei" bacteria --section genbank --formats fasta,gff --progress-bar --parallel 3 -o genbank
ncbi-genome-download --genera "Pseudomonas reinekei" bacteria --section refseq --formats fasta,gff --progress-bar --parallel 3 -o refseq
## 根据属名
ncbi-genome-download --genera Pseudomonas bacteria
## 物种名或属名可以放txt中
ncbi-genome-download --genera ref.txt bacteria
0.创建文件夹
mkdir summary/data/subset -p
for i in {UB002};do echo $i;touch $i.txt;mkdir $i;done
1.截取250万条reads
zcat xx路径/13_S52_L002_R1_001.fastq.gz |head -5000000 > summary/data/subset/13_S52_L002_R1_001.fq
## for循环
for i in {UB002};do for j in {1,2} ; do zcat 分发路径/2.clean_data/$i/${i}_S*_L007_R${j}_001.fastq.gz |head -5000000 > summary/data/subset/${i}_L007_R${j}_001.fq & done; done
2.下载参考基因组
## 把样品对应的属名物种名放进样品txt中
mamba activate ncbi_genome_download
## for循环 每个文件夹下有对应txt
for i in {UB002};do mv $i.txt $i; done
--- refseq数据库
for i in {UB002};do cd $i && ncbi-genome-download --genera $i.txt bacteria --formats fasta,gff --progress-bar --parallel 3 & done
for i in P{10P11,16P17,5}; do cd $i && ncbi-genome-download --genera *.txt fungi --formats fasta,gff --progress-bar --parallel 3 & done
--- genbank数据库
for i in P{10P11,16P17,5}; do cd $i && ncbi-genome-download --genera *.txt fungi --formats fasta,gff --progress-bar --parallel 3 --section genbank -o ./ & done
3.比对运行
cd UB002 && perl /share/nas2/yuj/project/2024/re-sequencing/GP-20240220-7911_20240509/pipline/bwa2pip.pl -i 项目路径/summary/data/subset -g refseq/bacteria -o ident -p UB002 && cd - &
## for循环
for i in {UB0029};do rm $i/ident -rf ;done
for i in {UB0029};do echo $i; cd $i && perl /share/nas2/yuj/project/2024/re-sequencing/GP-20240220-7911_20240509/pipline/bwa2pip.pl -i 项目路径/summary/data/subset -g */fungi -o ident -p $i && cd - & done
for i in {UB0029};do echo $i; cd *$i* && perl /share/nas2/yuj/project/2024/re-sequencing/GP-20240220-7911_20240509/pipline/bwa2pip.pl -i 项目路径/summary/data/subset -g */fungi -o $i.ident -p $i && cd - & done # 不同样品的参考放一块了,需要分别对样品进行比对
# summary/data/subset里面是每个样品截取后的序列
# refseq/bacteria文件夹下有很多以登录号命名的文件夹
# */fungi文件夹下有很多以登录号命名的文件夹
4.查看结果
for i in {UB002};do head -n 1 $i/ident/map.stat.xls;done | sort -k 3 -n > ref_sort.txt
cat ref_sort.txt
## 看看有没有gff
后n个都是比对大于70%的
n=11 && for i in $(cat ref_sort.txt | tail -n $n | cut -f 2);do ll */refseq/bacteria/$i/*.gff*;done
5.没有参考基因组的,尝试genbank数据库
mamba activate ncbi_genome_download
n=11 && for i in $(cat ref_sort.txt | head -n $n | cut -f 1);do cd $i && ncbi-genome-download --genera $i.txt bacteria --section genbank --formats fasta,gff --progress-bar --parallel 3 -o ./ & done
for i in $(cat ref_sort.txt | head -n $n | cut -f 1);do echo $i; cd $i && perl /share/nas2/yuj/project/2024/re-sequencing/GP-20240220-7911_20240509/pipline/bwa2pip.pl -i 项目路径/summary/data/subset -g genbank/genbank/bacteria -o ident2 -p $i && cd - & done
for i in $(cat ref_sort.txt | head -n $n | cut -f 1);do head -n 1 $i/ident2/map.stat.xls;done | sort -k 3 -n > ref_sort2.txt
cat ref_sort2.txt
n=11 && for i in $(cat ref_sort2.txt | tail -n $n | cut -f 2);do ll */genbank/genbank/bacteria/$i/*.gff*;done # 后n个是比对大于70%的
while read -r line; do echo "$line"; cd $(echo "$line" | cut -f 1) && ln -s refseq/bacteria/$(echo "$line" | cut -f 2) data;cd -; done < 11ok.txt
for i in $(cat 11ok.txt | cut -f 1);do cd $i && gunzip data/*.gz && cd data && ir.py -i *.fna -l | sort -k 1 -nr > len.txt && cd - & done
for i in $(cat 11ok.txt | cut -f 1);do cat $i/data/len.txt ;done
共进行了5轮基因组下载比对
1.(一轮比对)根据第1次菌种鉴定结果,
对每个样品利用ncbi-genome-download v0.3.3(https://github.com/kblin/ncbi-genome-download)从refseq数据库下载对应鉴定物种们的基因组,
从测序数据中抽取125万条reads后使用bwa-mem2 v2.2.1(https://github.com/bwa-mem2/bwa-mem2)将reads比对参考基因组,
对获得的sam文件使用samtools v1.9(https://github.com/samtools/samtools/)软件的flagstat功能进行统计获得匹配率,
从匹配率大于70%的众多基因组中挑选比对率最高的基因组作为参考。
2.(二轮比对)若某个样品没有匹配率大于70%的基因组,
再使用ncbi-genome-download从genbank数据库下载对应物种们的基因组,
重新抽取reads比对统计,从匹配率大于70%的众多基因组中挑选比对率最高的基因组作为参考。
3.若某个样品依旧没有大于70的参考,
则对该样品使用spades v3.15.5(https://github.com/ablab/spades)进行初步组装,
从组装结果中挑选100k左右的contig到ncbi使用blast进行比对获得潜在的鉴定物种,取前5个物种。
4.(三轮比对)将第3步的潜在物种们和第2次提供的菌种鉴定结果整合,
重复1步骤
5.(四轮比对)若某个样品没有匹配率大于70%的基因组,
重复2步骤
6.(五轮比对)若在第4、第5步中对某个样品操作时,遇到基因组多到软件下载不下来中途报错的情况,
则根据第4步的整合结果,将该样品对应的潜在物种们放宽至属水平,把其他样品下载下来的同属基因组整合后,
进行比对统计,挑选大于70%的参考基因组
7.若依旧没有,就没有其他操作,定为找不到
9️⃣.其他
----报告内容
变异在基因组上的分布--circos软件展示
## 3.7 BSA关联分析
### 3.7.3 关联区域内基因注释
GO
COG
通路代谢
KEGG
KEGG富集分析--散点图
----对应软件
数据质控--fastp
比对参考基因组--bwa 输出sam文件
转换--samtools 输出bam文件
gatk_haplotypecaller--变异位点
gatk_VariantFiltration--过滤
snpeff--注释变异(SNP、Small InDel)和预测变异影响
breakdance--sv结构变异检测
vcftools--获取可信变异位点
python脚本&ggplot2--混池SNPindex数据拟合&绘图
blastall--区间内基因功能注释
primer3--设计区间内indel标记的引物
----分析流程
step7 过滤后获取可信变异位点
step8 sv结构变异检测
step9 区间内基因注释