Vcftools及vcf
一. VCF文件
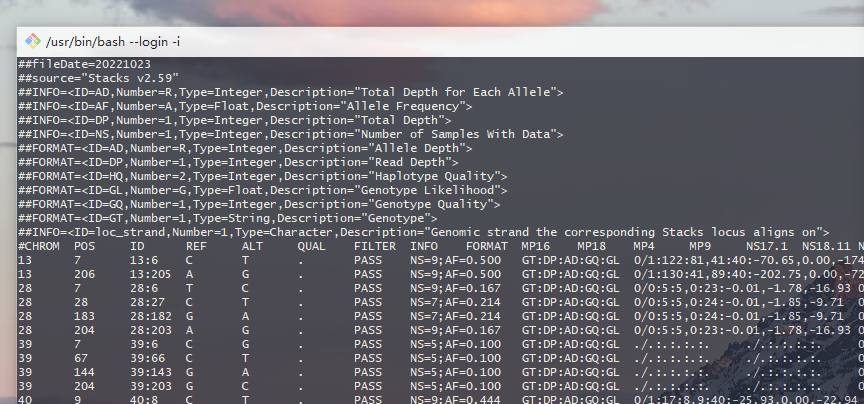
1.1 简介
以“#”开头的注释部分:VCF的介绍信息,通常以##作为起始,其后一般接以FILTER,INFO,FORMAT等字样。
以##FILTER开头的行,表示注释VCF文件当中第7列中缩写词的说明,比如q10为Quality below 10;##INFO开头的行注释VCF第8列中的缩写字母说明,比如AF代表Allele Frequency也就是等位基因频率;##FILTER开头的行注释VCF第9列中的缩写字母说明;另外还有其他的一些信息,文件版本"fileformat=VCFv4.0"等等。
没有“#”开头的主体部分:每一行代表一个variant,各列之间用tab空白隔开,前面9列为固定列,第10列开始为样品信息列,可以无限多个
# 主体部分10列的范例
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT MP16
13 7 13:6 C T . PASS NS=9;AF=0.500 GT:DP:AD:GQ:GL 0/1:122:81,41:40:-70.65,0.00,-174.69
1.2 10列分别代表的意义
1.CHROM:染色体编号
2.POS:variant所在的left-most位置(1-base position,发生变异的位置的第一个碱基所在的位置)
3.ID:variant的ID,对应着dbSNP数据库中的ID。(SNP/INDEL的dbSNP编号通常以rs开头,一般只有人类基因组才有dbSNP编号,若没有,则默认使用‘.’)
4.REF:参考序列的Allele。(等位碱基,即参考序列该位置的碱基类型及碱基数量,必须是A,C,G,T,N且都大写)
5.ALT:variant的Allele,若有多个,则使用逗号分隔。(变异的碱基类型及碱基数量)这里的碱基类型和碱基数量,对于SNP来说是单个碱基类型的编号,而对于Indel来说是指碱基个数的添加或缺失,以及碱基类型的变化
6.QUAL:variants的质量。PPhred格式(Phred_scaled)的质量值,代表着此位点是纯合的概率,此值越大,则纯合概率越低,表示突变可能性越大。(表示变异碱基的可能性)
计算方法:Phred值 = -10 * log (1-p), p为variant存在的概率; 通过计算公式可以看出值为10的表示错误概率为0.1,该位点为variant的概率为90%。
7.FILTER:使用上一个QUAL值来进行过滤的话,是不够的。GATK能使用其它的方法来进行过滤,过滤结果中通过则该值为”PASS”;若variant不可靠,则该项不为”PASS”或为”.”。
8.INFO:variant的相关信息
9.FORMAT:variants的格式,例如GT:AD:DP:GQ:PL
10.SAMPLES:各个Sample的值,由BAM文件中的@RG下的SM标签所决定,这些值对应着第9列的各个格式,不同格式的值用冒号分开,每一个sample对应着1列;多个samples则对应着多列,这种情况下列的数多余10列。
1.3 FORMAT 及对应 SAMPLES 值说明
FORMAT字段与样本数据列共同构成基因型信息,格式为 FORMAT字段:样本数据。例如:
GT:AD:DP:GQ:PL 0/1:6,5:11:99:138,0,153
1. GT(GenoType)
格式特征:
- 使用
|或/分隔两个数字(二倍体) - 单倍体(如叶绿体基因组)直接显示单一数字
|表示 Phased genotype(明确等位基因来源的染色体)/表示 Unphased genotype(未明确染色体来源)
数值含义:
| 基因型 | 二倍体含义 | 单倍体含义 |
|---|---|---|
0 |
- | 与REF一致 |
1 |
- | 与ALT一致 |
0/0 |
纯合REF | 不适用 |
0/1 |
杂合REF/ALT | 不适用 |
1/1 |
纯合ALT | 不适用 |
./. |
数据缺失 | 数据缺失 |
2. AD(Allele Depth)
数据格式:逗号分隔的数值
含义:各等位基因的reads覆盖深度
二倍体示例:6,5 → REF深度6,ALT深度5
3. DP(Depth)
计算方式:DP = SUM(AD)
单倍体特殊性:可能直接表示唯一等位基因深度
4. GQ(Genotype Quality)
特性:
- Phred格式质量值(
GQ = -10 * log10(1-P),P为基因型正确概率) - 单倍体与二倍体计算规则相同
5. PL(Genotype Likelihoods)
数据格式:
- 二倍体:逗号分隔的三个值(对应
0/0, 0/1, 1/1) - 单倍体:逗号分隔的两个值(对应
0, 1)
计算规则:PL = -10 * log10(p)(p为对应基因型概率)
1.4 INFO 信息列
AC=1;AF=0.500;AN=2;BaseQRankSum=0.748;ClippingRankSum=0.000;DB;DP=34;ExcessHet=3.0103;FS=3.424;MLEAC=1;MLEAF=0.500;MQ=31.07;MQRankSum=-0.087;QD=11.87;ReadPosRankSum=-1.349;SOR=2.636
AC=2;AF=1.00;AN=2;DB;DP=14;ExcessHet=3.0103;FS=0.000;MLEAC=2;MLEAF=1.00;MQ=31.60;QD=29.36;SOR=5.421
| TAG | 描述 | 示例值 | 备注 |
|---|---|---|---|
| AC | 等位基因计数(Allele Count),与变异一致的Allele总数 | AC=1, AC=2 | 双倍体样本中,0/1基因型对应AC=1,1/1对应AC=2 |
| AF | 等位基因频率(Allele Frequency),AF = AC / AN | AF=0.500, AF=1.00 | 双倍体样本中,0/1基因型对应AF=0.5,1/1对应AF=1.0 |
| AN | 总等位基因数(Allele Number) | AN=2 | 双倍体样本固定为2 |
| BaseQRankSum | Alt与Ref碱基质量的Wilcoxon秩和检验Z值 | 0.748 | 负值表示Alt碱基质量低于Ref |
| ClippingRankSum | Alt与Ref硬剪切碱基数的Wilcoxon秩和检验Z值 | 0.000 | 用于检测剪切偏好性 |
| DB | 标记该变异位点存在于公共数据库中 | 无具体值 | 仅存在标记(如"DB")表示True |
| DP | 过滤后reads覆盖深度 | DP=34, DP=14 | 区别于未过滤的原始DP |
| ExcessHet | 过量杂合性的Phred标准化p值 | 3.0103 | 值越大越可能错误 |
| FS | Fisher精确检验的链偏好性Phred值 | 3.424, 0.000 | 值越大链偏好性越显著,推荐保留FS < 10-20 |
| MLEAC | 最大似然估计的等位基因计数 | MLEAC=1, MLEAC=2 | 可能与AC不同 |
| MLEAF | 最大似然估计的等位基因频率 | MLEAF=0.500 | 可能与AF不同 |
| MQ | 比对质量的均方根值(RMS Mapping Quality) | 31.07, 31.60 | 值越高比对质量越好 |
| MQRankSum | Alt与Ref比对质量的Wilcoxon秩和检验Z值 | -0.087 | 负值表示Alt比对质量差于Ref,推荐保留 > -1.65~-3.0 |
| QD | 深度标准化变异质量(Variant Confidence/Quality by Depth) | 11.87, 29.36 | 值越高可信度越高 |
| ReadPosRankSum | Alt与Ref读段位置的Wilcoxon秩和检验Z值 | -1.349 | 负值表示Alt更靠近reads末端,推荐保留 > -1.65~-3.0 |
| SOR | 链偏倚对称比值比(Symmetric Odds Ratio) | 2.636, 5.421 | 值越大链偏好性越强(>3表示可能存在偏好性) |
| Dels | 含跨缺失reads的比例 | 未显示示例 | 值为0表示SNP,无此TAG表示INDEL |
| HaplotypeScore | 与最多两个分离单倍型的一致性 | 未显示示例 | 值越高表示单倍型不一致风险越高 |
| InbreedingCoeff | 基于哈迪-温伯格平衡的近交系数 | 未显示示例 | 用于检测群体结构异常 |
| MQ0 | 比对质量为零的reads总数 | 未显示示例 | 值高可能表示比对问题 |
| RPA | 串联重复单元的重复次数(每个等位基因) | 未显示示例 | 与STR相关 |
| RU | 串联重复单元序列 | 未显示示例 | 例如"RU=GAA"表示重复单元为GAA |
| STR | 标记短串联重复变异 | 未显示示例 | 存在标记表示该变异为短串联重复 |
关键过滤建议:
- FS:推荐保留 < 10-20
- MQRankSum:推荐保留 > -1.65 ~ -3.0
- ReadPosRankSum:推荐保留 > -1.65 ~ -3.0
- QD:通常过滤低QD值(如 < 2)
- SOR:> 3时可能存在链偏好性风险
二. Vcftools用法
2.1 过滤变异类型
vcf文件中可能会同时包含snp以及indel两种变异类型,vcftools可以很快的将两者进行分离。
2.1.1 过滤掉indel,只保留snp,用到的命令选项:–remove-indels。
vcftools --remove-indels --recode --recode-INFO-all --vcf raw.vcf --stdout >raw.snp.vcf
2.1.2 过滤掉snp,只保留indel,用到的命令选项:–keep-only-indels。
vcftools --keep-only-indels --recode --recode-INFO-all --vcf raw.vcf --stdout >raw.indel.vcf
这样,就可以分别得到只包含snp和indel的vcf文件。
2.2 筛选指定位置变异位点
vcftools还可以挑选出基因组上某些区域的变异信息。
vcftools --vcf Variants.snp.unknown_multianno.vcf --chr A03 --from-bp 577700 --to-bp 607700 --out out_prefix --recode --recode-INFO-all
这里解释一下各个参数:
–vcf:后面跟的是vcf文件
–chr:后面跟筛选区域所在的染色体
–form-bp:后跟筛选区域的起始位置
–to-bp:后跟筛选区域的终止位置
–out:输出文件的前缀
–recode:没有此参数则不会输出
2.3 过滤指定缺失率的变异位点
vcf 文件中很多snp在某些样品中是缺失的,也就是基因型为 “./.” 。如果缺失率较高,这种snp位点在很多分析中是不能用的,需要去掉。这里用到的选项是 --max-missing。
vcftools --vcf snp.vcf --recode --recode-INFO-all --stdout --max-missing 1 > snp.new.vcf
--max-missing 后跟的值为 0-1 ,1代表不允许缺失,0代表允许全部缺失。
2.4 计算snp缺失率
vcftools中有两个参数可以计算vcf文件中snp的缺失率。
分别是:
–missing-indv:生成一个文件,报告每个样品的缺失情况,该文件的后缀为“.imiss”。
–missing-site:生成一个文件,报告每个snp位点的缺失情况,该文件的后缀为“.lmiss”。
2.4.1 –missing-site
vcftools --vcf snp.vcf. --missing-site
运行以上命令后会在当前目录生成一个 out.lmiss 文件,其格式如下:
CHR POS N_DATA N_GENOTYPE_FILTERED N_MISS F_MISS
chr01 194921 988 0 368 0.37247
chr01 384714 988 0 204 0.206478
chr01 384719 988 0 202 0.204453
chr01 518438 988 0 488 0.493927
chr01 518473 988 0 452 0.45749
chr01 518579 988 0 418 0.423077
chr01 518635 988 0 428 0.433198
chr01 680786 988 0 346 0.350202
chr01 680834 988 0 412 0.417004
# 前两列为snp所在位置,第三列为等位基因总数,第5列为缺失的总数,最后一列为缺失率。
2.4.2 –missing-indv
vcftools --vcf snp.vcf. --missing-indv
运行以上命令后会在当前目录生成一个 out.imiss 文件,其格式如下:
INDV N_DATA N_GENOTYPES_FILTERED N_MISS F_MISS
1 8747 0 3632 0.415228
10 8747 0 1264 0.144507
102 8747 0 2016 0.230479
105 8747 0 6322 0.722762
106 8747 0 2365 0.270378
107 8747 0 4376 0.500286
108 8747 0 5682 0.649594
109 8747 0 1877 0.214588
11 8747 0 1039 0.118784
# 第一列为样品名称,第二列为总的snp数,第4列为缺失的总数,最后一列为缺失率。
2.5 随机抽取指定个样品
vcftools可以随机抽取指定个样品的vcf文件,用到的选项为 --max-indv ,指定要从vcf文件中随机抽取指定个样品。
# 随机抽取5个样品,执行以下代码:
vcftools --vcf snp.vcf --max-indv 5 --remove-indels --recode --out outfilename